Fapesp
- Política

Contra cortes na Fapesp, APqc cita pesquisa que ajudou a sequenciar o Coronavírus
Audiência Pública discutiu proposta que pode cortar 30% da Fapesp
- Política

Audiência discute risco de cortes na Fapesp
Audiência pública ocorre nesta terça-feira (21), na Alesp
- Saúde

Startup cria teste para rastrear câncer de colo do útero
Projeto é apoiado pela Fapesp
- Covid-19

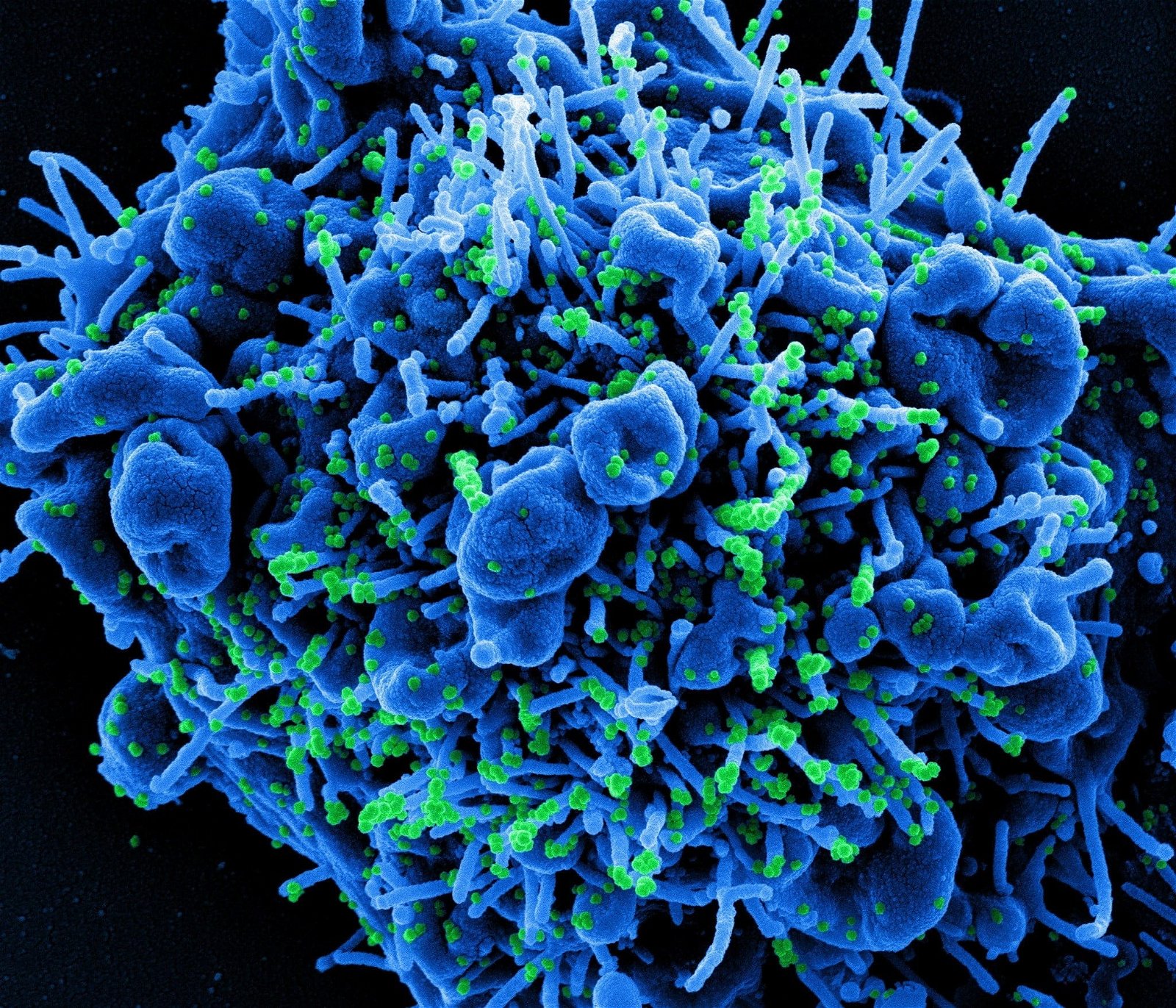
Descoberta de anticorpos pode ajudar no tratamento da covid-19
Anticorpos associados a doenças autoimunes pode sinalizar a gravidade da covid-19
- Capital

Sistema vai monitorar variantes da covid-19 na Capital
Meta é analisar semanalmente 384 amostras de pacientes para monitorar variantes
- Covid-19

Cientistas investigam pessoas imunes à covid-19
Estudo analisou amostras de 86 casais, sendo que um dos parceiros não se contaminou
- Saúde

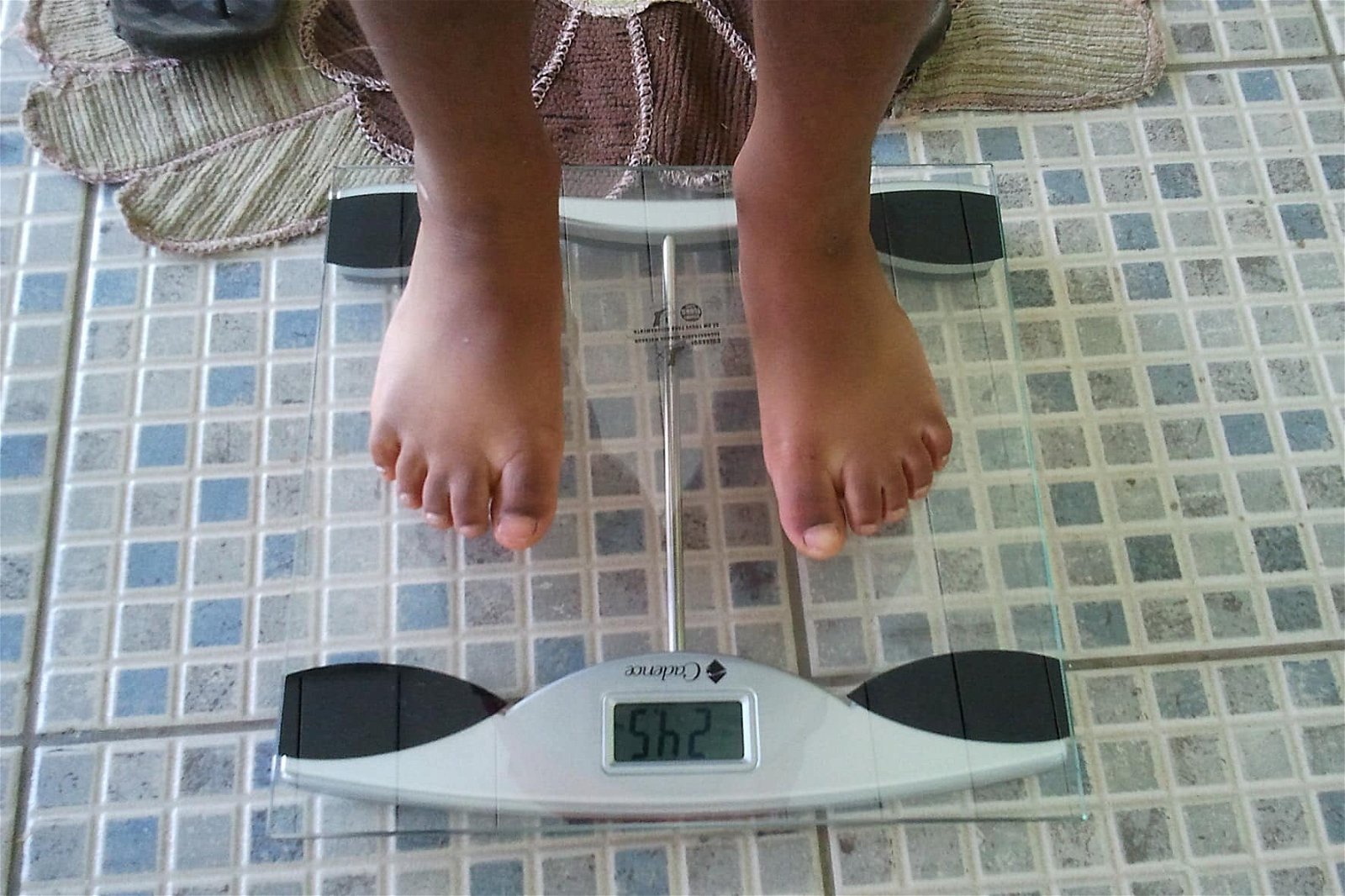
Meninas obesas têm mais chances de doenças cardiovasculares na vida adulta
Estudo de cientistas brasileiros comparou meninos e meninas
- Covid-19

Eficiência das máscaras varia entre 15% e 98%, dependendo do tecido
Estudo da USP testou 227 modelos de máscaras
- Covid-19

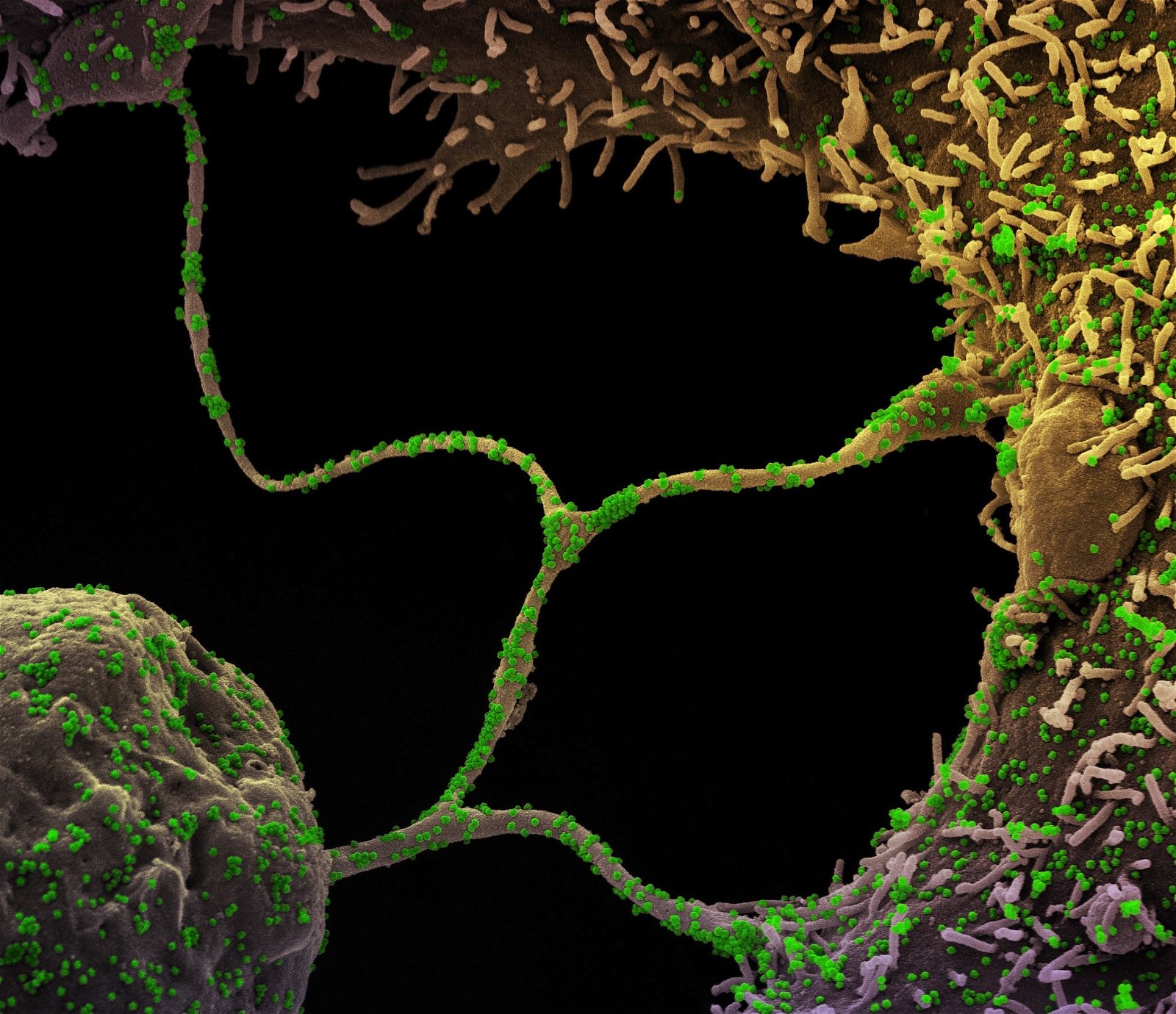
Cientistas encontram coronavírus na gengiva de pacientes
Estudo contribui para desvendar uma das possíveis fontes do novo coronavírus na saliva
- Covid-19

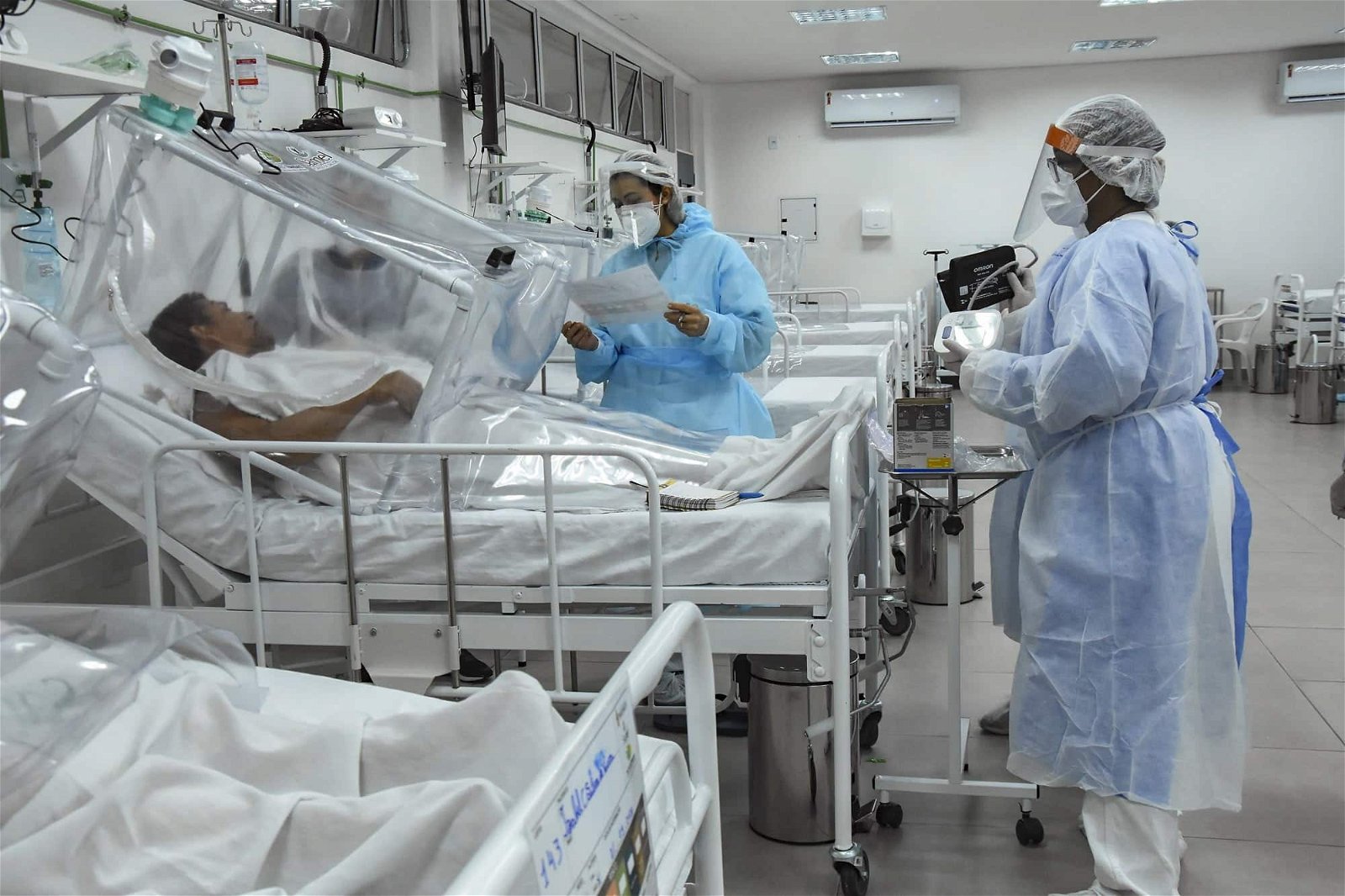
Variante brasileira é mais transmissível e pode levar a reinfecção, sugere estudo
Em sete semanas, variante se tornou a mais prevalente em Manaus